
HelixFold-Single 模型
HelixFold-Single
HelixFold-Single类别文心-生物计算应用蛋白结构预测模型概述秒级别的蛋白结构预测模型,是业界首个开源的基于单序列语言模型的蛋白结构预测大模型,从近3亿的无标注蛋白质数据中隐式的学习MSA信息,在90% 的单体蛋白场景上预测效果持平AF2。在抗体结构预测场景下,比AlphaFold2预测结果更优。模型说明模型说明模型简介通过人工智能的方法,从蛋白质的一级序列精准预测
-
*.2折
官方价格:咨询
-
*.2折
官方价格:咨询
-
*.2折
官方价格:咨询
HelixFold-Single
类别文心-生物计算
应用蛋白结构预测
模型概述
秒级别的蛋白结构预测模型,是业界首个开源的基于单序列语言模型的蛋白结构预测大模型,从近3亿的无标注蛋白质数据中隐式的学习MSA信息,在90% 的单体蛋白场景上预测效果持平AF2。在抗体结构预测场景下,比AlphaFold2预测结果更优。
模型说明
模型说明
模型简介
通过人工智能的方法,从蛋白质的一级序列精准预测其三维结构,已被AlphaFold2证实可达到实验精度。但AlphaFold2的模型主要依赖多序列比对(MSA)和模版(Template)信息,而从蛋白质数据库中搜索MSA和模版又是一件非常耗时的工作,成为模型向产业界大规模推广的一个瓶颈。百度飞桨螺旋桨与百图生科,共同开发了新的蛋白结构预测大模型,不需要MSA信息作为输入,仅仅通过蛋白质的一级序列就可以准确预测其三级结构。该模型已经正式在螺旋桨PaddleHelix平台对外开源,并提供web服务在线测试体验。未来,该模型还将作为百图生科xTrimo多模态生物计算大模型体系的重要组成部分,与其他蛋白质、相互作用、细胞模型一起,助力百图生科的创新蛋白质药物研发。
原理介绍
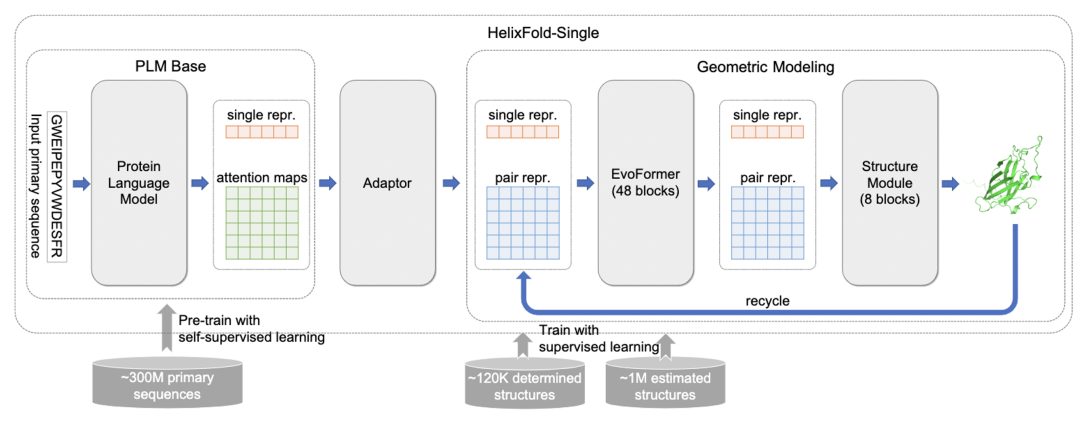
HelixFold-Single将大规模蛋白质语言模型与 AlphaFold2 的卓越几何学习能力相结合。它首先利用自我监督学习范式预训练了具有数以百万计的初级序列的大规模蛋白质语言模型 (PLM),该范式将用作 MSA 和学习模板的替代方案协同进化信息。然后,我们通过将预训练的 PLM 和 AlphaFold2 的基本组件相结合,获得了一个端到端的,可仅从以及序列出发预测院子三维坐标的可微模型。
使用方法
下载训好的模型
wget https://baidu-nlp.bj.bcebos.com/PaddleHelix/HelixFold-Single/helixfold-single.pdparams
进行预测
python helixfold_single_inference.py \–init_model=./helixfold-single.pdparams \
–fasta_file=data/7O9F_B.fasta \
–output_dir="./output"
详见github文档
模型效果
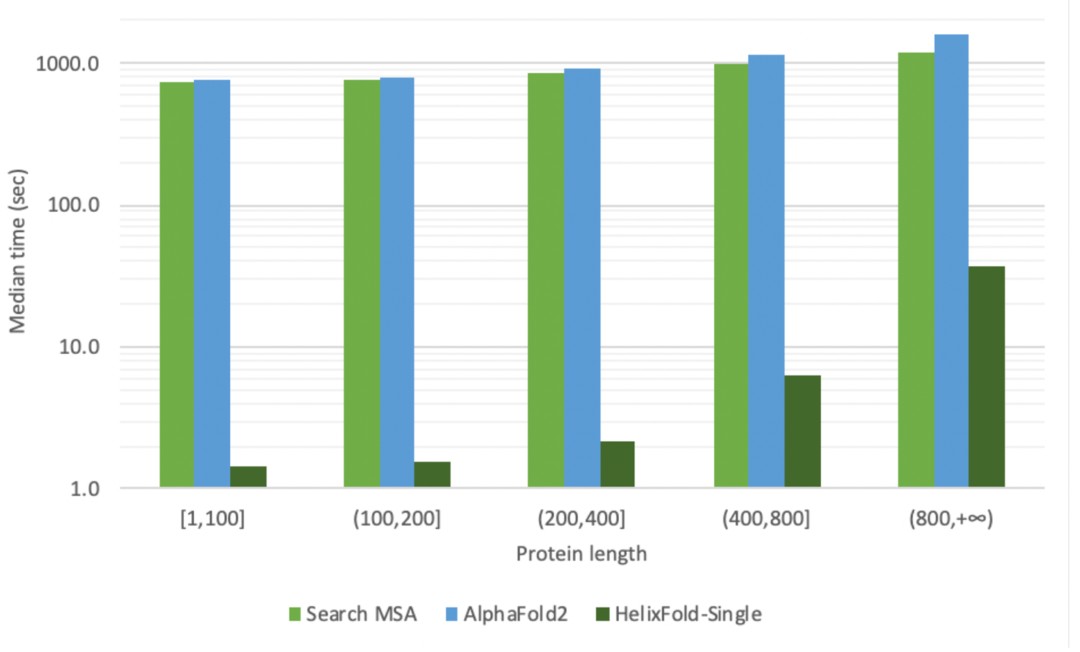
相较AlphaFold2, HelixFold-Single模型推理的速度平均提升数百倍。

新闻资讯
关注人脸、文字识别、关注ai智能未来- 百度离线活体检测精度怎么样 2024-01-17
- 百度云ocr私有化部署是如何计费的 2024-01-17
- 百度智能云AI作画高级版API全方面升级 2024-01-17
- 房产证OCR识别算法,快来免费试用! 2024-01-17
- 曦灵数字人平台重磅升级,限时邀测 2024-01-17
- 百度云文档解析api接口 2024-01-17

